
Fragment notatki:
Wykład 5
16.03
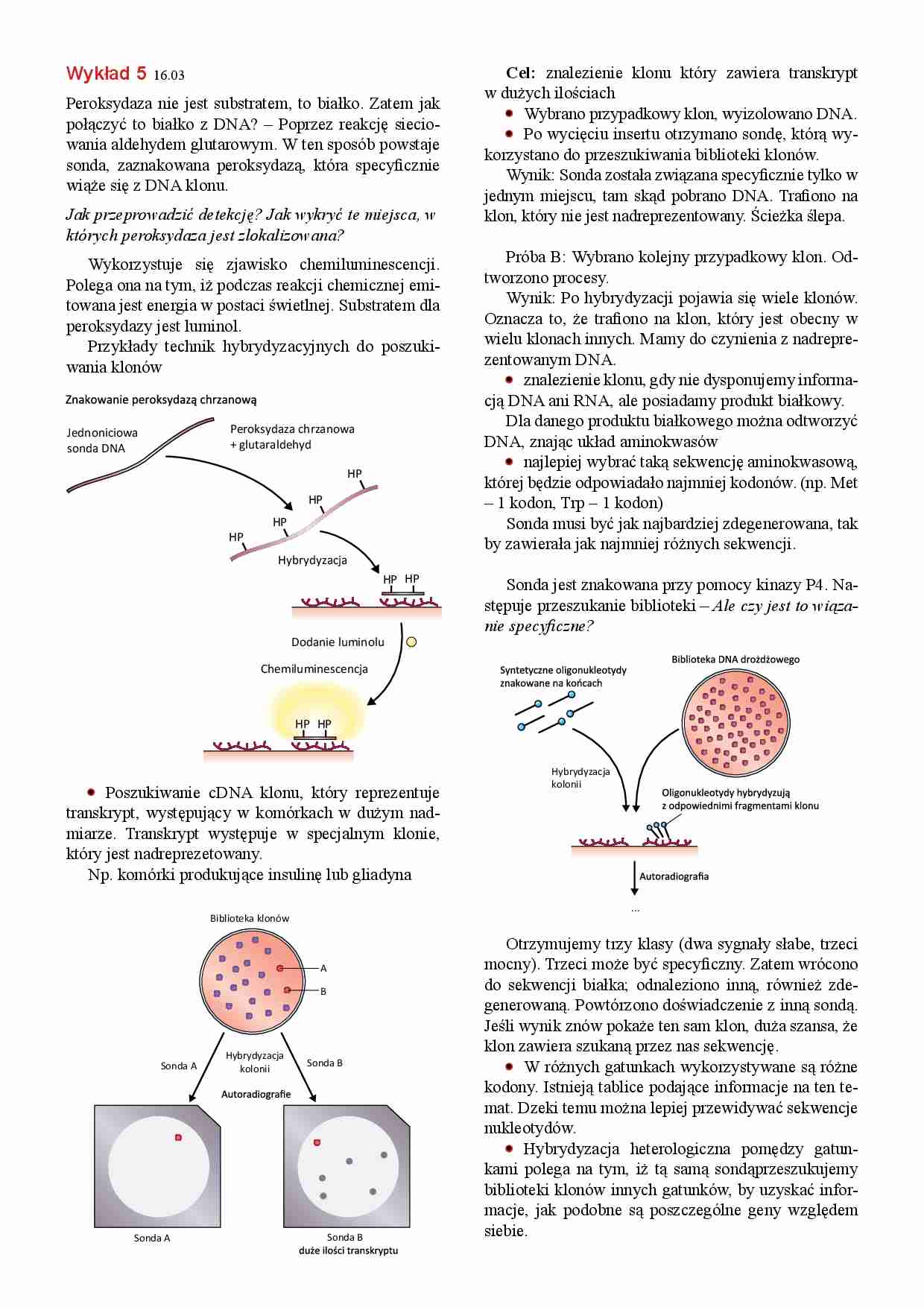
Peroksydaza nie jest substratem, to białko. Zatem jak
połączyć to białko z DNA? – Poprzez reakcję sieciowania aldehydem glutarowym. W ten sposób powstaje
sonda, zaznakowana peroksydazą, która specyficznie
wiąże się z DNA klonu.
Jak przeprowadzić detekcję? Jak wykryć te miejsca, w
których peroksydaza jest zlokalizowana?
Wykorzystuje się zjawisko chemiluminescencji.
Polega ona na tym, iż podczas reakcji chemicznej emitowana jest energia w postaci świetlnej. Substratem dla
peroksydazy jest luminol.
Przykłady technik hybrydyzacyjnych do poszukiwania klonów
Peroksydaza chrzanowa
+ glutaraldehyd
Jednoniciowa
sonda DNA
HP
HP
HP
HP
Hybrydyzacja
HP HP
Dodanie luminolu
Cel: znalezienie klonu który zawiera transkrypt
w dużych ilościach
• Wybrano przypadkowy klon, wyizolowano DNA.
• Po wycięciu insertu otrzymano sondę, którą wykorzystano do przeszukiwania biblioteki klonów.
Wynik: Sonda została związana specyficznie tylko w
jednym miejscu, tam skąd pobrano DNA. Trafiono na
klon, który nie jest nadreprezentowany. Ścieżka ślepa.
Próba B: Wybrano kolejny przypadkowy klon. Odtworzono procesy.
Wynik: Po hybrydyzacji pojawia się wiele klonów.
Oznacza to, że trafiono na klon, który jest obecny w
wielu klonach innych. Mamy do czynienia z nadreprezentowanym DNA.
• znalezienie klonu, gdy nie dysponujemy informacją DNA ani RNA, ale posiadamy produkt białkowy.
Dla danego produktu białkowego można odtworzyć
DNA, znając układ aminokwasów
• najlepiej wybrać taką sekwencję aminokwasową,
której będzie odpowiadało najmniej kodonów. (np. Met
– 1 kodon, Trp – 1 kodon)
Sonda musi być jak najbardziej zdegenerowana, tak
by zawierała jak najmniej różnych sekwencji.
Sonda jest znakowana przy pomocy kinazy P4. Następuje przeszukanie biblioteki – Ale czy jest to wiązanie specyficzne?
Chemiluminescencja
HP HP
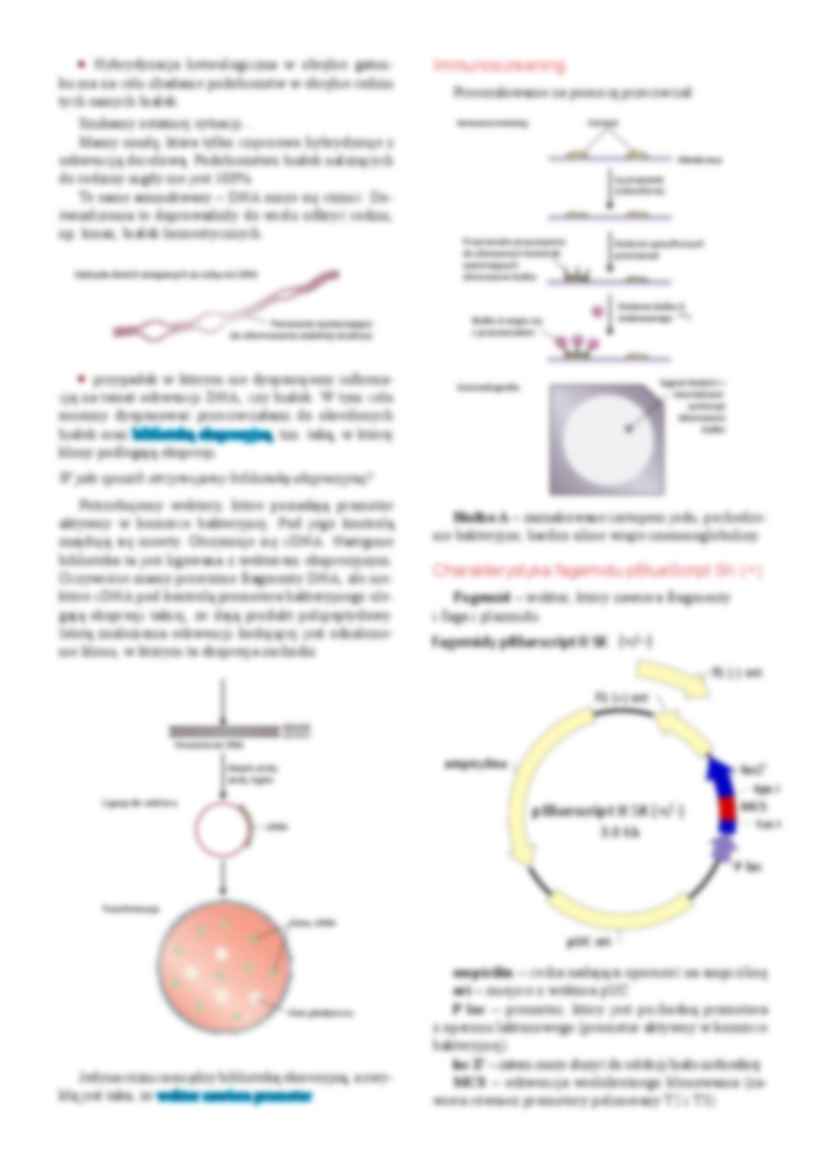
• Poszukiwanie cDNA klonu, który reprezentuje
transkrypt, występujący w komórkach w dużym nadmiarze. Transkrypt występuje w specjalnym klonie,
który jest nadreprezetowany.
Np. komórki produkujące insulinę lub gliadyna
...
Biblioteka klonów
A
B
Sonda A
Sonda A
Hybrydyzacja
kolonii
Hybrydyzacja
kolonii
Sonda B
Sonda B
Otrzymujemy trzy klasy (dwa sygnały słabe, trzeci
mocny). Trzeci może być specyficzny. Zatem wrócono
do sekwencji białka; odnaleziono inną, również zdegenerowaną. Powtórzono doświadczenie z inną sondą.
Jeśli wynik znów pokaże ten sam klon, duża szansa, że
klon zawiera szukaną przez nas sekwencję.
• W różnych gatunkach wykorzystywane są różne
kodony. Istnieją tablice podające informacje na ten temat. Dzeki temu można lepiej przewidywać sekwencje
nukleotydów.
• Hybrydyzacja heterologiczna pomędzy gatunkami polega na tym, iż tą samą sondąprzeszukujemy
biblioteki klonów innych gatunków, by uzyskać informacje, jak podobne są poszczególne geny względem
siebie.
• Hybrydyzacja heterologiczna w obrębie gatunku ma na celu zbadanie podobieństw w obrębie rodzin
tych samych białek.
Szukamy ostatniej sytuacji...
Mamy sondę, która tylko częsciowo hybrydyzuje z
sekwencją docelową. Podobieństwo białek należących
do rodziny nigdy nie jest 100%.
Te same aminokwasy – DNA może się różnić. Doświadczenia te doprowadziły do wielu odkryć rodzin,
np. kinaz, białek homeotycznych.
Immunoscreening
Przeszukiwanie za pomocą przeciwciał:
Immunoscreening
Kolonie
Membrana
Liza komórek
(chloroform)
125
I
• przypadek w którym nie dysponujemy informacją na temat sekwencji DNA, czy białek. W tym celu
musimy dysponować przeciwciałami do określonych
białek oraz biblioteką ekspresyjną, tzn. taką, w której
klony podlegają ekspresji.
W jaki sposób otrzymujemy bibliotekę ekspresyjną?
Potrzebujemy wektory, które posiadają promotor
aktywny w komórce bakteryjnej. Pod jego kontrolą
znajdują się inserty. Otrzymuje się cDNA. Następnie
biblioteka ta jest ligowana z wektorem ekspresyjnym.
Oczywiście mamy przeróżne fragmenty DNA, ale niektóre cDNA pod kontrolą promotora bakteryjnego ulegają ekspresji takiej, że dają produkt polipeptydowy.
Istotą znalezienia sekwencji kodującej jest odnalezienie klonu, w którym ta ekspresja zachodzi
Białko A – zaznakowane izotopem jodu, pochodzenie bakteryjne, bardzo silnie wiąże immunoglobuliny.
Charakterystyka fagemidu pBlueScript SK (+)
Fagemid – wektor, który zawiera fragmenty
i faga i plazmidu.
Fagemidy pBluescript II SK (+/–)
f1 (-) ori
f1 (+) ori
AAAAA
TTTTT
Dwuniciowe DNA
ampicylina
lacZ'
ends, ligate
Kpn I
Ligacja do wektora
pBluescript II SK (+/-)
cDNA
3.0 kb
MCS
Sac I
P lac
Transformacja
klony cDNA
pUC ori
klon gliadynowy
Jedyna różnica między biblioteką eksresyjną, a zwykłą jest taka, że wektor zawiera promotor.
ampicilin – cecha nadająca oporność na ampicilinę
ori – miejsce z wektora pUC
P lac – promotor, który jest pochodną promotora
z operonu laktozowego (promotor aktywny w komórce
bakteryjnej)
lac Z’ – zatem może służyć do selekcji biało niebieskiej
MCS – sekwencja wielokrotnego klonowania (zawiera również promotory polimerazy T2 i T3).
SacI, Kpn1 – miejsca restrykcyjne
f1(+) ori – fragment z faga f1 (działanie: możemy
cały czas używać fagemidu jako zwykłego wektora,
a po zakażeniu komórki będzie wykorzystywana sekwencja f1(+) ori i od tej sekwencji zachodzić będzie
replikacja, która zakończy kolistą cząsteczkę DNA jedną z nici wektora).
Wersja (-) przy odwróconej sekwencji f1(+) ori.
Funkcje:
• klonowanie fragmentów DNA
• separacja molekularna
• zwielokrotnienie ilości
• otrzymanie w postaci pojedynczej nici
• wprowadzenie nadekspresji białka
• selekcja biało-niebieska
• otrzymanie transkryptów in-vitro
Reakcja pCR
Reakcja syntezy DNA. Cechą szczególną jest to, że
syteza (w bardzo szczególnych warunkach) jest powtarzana wielokrotnie. Następuje amplifikacja sekwencji
DNA.
Przed wynalezieniem metody PCR jedynym sposobem otrzymania DNA było klonowanie (wbudowanie
insertu do wektora i podział komórki).
Do reakcji PCR potrzebujemy:
• startery
• matrycę DNA
• polimerazę
Możliwe jest hybyrydyzowanie ze sobą cząsteczek
pierwonych matrycy, ale preferowane jest hybrydyzowanie ze starterem. Startery są zawsze w nadmiarze, są
to krótsze fragmenty, ddlatego są one preferowane.
3 cykl: Hybrydyzująze sobą produkty cyklu drugiego, które mają już określoną długość (w ten sposób
określana jest ostateczna długość produktów PCR).
Zwykle produkty PCR analizujemy przy pomocy
elektroforezy żelowej.
Co wpływa na powodzenie eksperymentu?
Przede wszystkim odpowiednie zaprojektowanie
starterów.
Startery są projektowane w oparciu o znaną już sekwencę, okalają odcinek DNA, który ma być powielany.
Starter przedni – sekwencja nici górnej.
Starter tylni – sekwencja nici dolnej.
Starter musi być specyficzny! Co to znaczy?
W całym genomie starter powinien parować tylko z
jedną komplementarą sekwencją! Taki wynik jest uzyskiwany za pomocą:
• odpowiedniej długości startera (szacuje się, że
dla startera o długości 17 pz można znaleźć sekwencję,
która się powtórzy tylko raz)
• konkretne temperatury
Typowy profil temperaturowy
100
Opis reakcji
Skąd bierze sę amplifikacja?
Z powtarzalności cylki (głównie od cyklu 3) mamy
już matrycę o zdefiniowanej długości.
1 cykl: Produkty, które są syntezowane mają długość określoną przez czas syntezy.
2 cykl: Mamy nić matrycową i produkt 1, znów następuje hybrydyzacja. Powstaą produkty, które maą już
określoną długość.
90
80
Temperatura °C
• Matryca jest denaturowana. Otrzymujemy dwie
nici, do których są hydrolizowane startery. Startery określają produkt, jaki będzie po reakcji (Długość
DNA)
W PCR matrycą jest pojedyncza cząsteczka (nić)
DNA dostarczana przez denaturację cząsteczki DNA
(w komórce nić dostarczona przy udziale białek, m.in.
helikaz).
Próbka jest następnie ochłądzana (temperatura
znacznie niższa niż przy denaturacji). Wartość temperatury jest określana przez charakter starterów, które
łączą się z obiema niciami.
• Synteza prowadzona przez termostabilną polimerazę DNA na obu niciach. Temperatura powyżej 70°C
(około 74°C)
Krytycznym etapem dla osiągnięcia specyficzności
jest etap hybrydyzacji.
Denaturacja
(1 min)
(2 min)
70
60
50
40
0
1
2
3
4
Czas (minuty)
5
6
7
Należy zwrócić uwagę na temperaturę wiązania
starterów
3 sytuacje hipotetyczne
• Ekstremalne wartości pH wpływają na denaturację DNA
• Wysoka temperatura powoduje rozerwanie wiązań wodorowych i dysocjację DNA. Zatem przy zbyt
wysokiej temperaturze startery nie hybrydyzują.
• Temperatura jest za niska – starter hybrydyzuje
tylko prezz niektóre reszty. Są to słabe oddziaływania.
Zależy nam, aby wszystkie reszty hybrydyzowały.
Temperatura topnienia DNA – temperatura, w której połowa cząsteczek DNA jest rozdysocjowana (połowowa cząsteczek związana lub nie)
Jak to oszacować?
T m = (4 × [G + C ] ) + (2 × [A + T ] )°C
prawdziwe dla względnie krótkich długości DNA
Temperatura hybrydyzuje – o 1-2°C niższa od temperatury topnienia → konieczność wykorzystania polimeraz termostabilnych.
Proces należy powtarzać około trzydzieści razy
(teoretycznie powinno się uzyskać 2n-2 cząsteczek).
Produktu może być trochę mniej, ponieważ polimeraz traci swoją aktywność podczas wzrostu temperatury oraz substraty ulegają rozkładowi częściowemu.
mimo to ilość produktu DNA jest wystarczająca do
dalszych badań.
... zobacz całą notatkę

Komentarze użytkowników (0)