

Fragment notatki:
Techniki są jasne do państwa?
Jeżeli ktoś czegoś nie rozumie to zapraszam do zadawania pytań, jeżeli nie to przejdziemy do
innych problemów i innych metod analizy produktów ekspresji genów, bo o tym mówimy.
Taką grupę metod stanowią metody w których wykorzystujemy mutacje, a wiec kilka słów na
temat mutagenezy. Mutageneza czy też mutacje, są czymś naturalnym. Wszyscy jak tu
siedzimy, jesteśmy podlegamy temu zjawisku. Mutacje zachodzą cały czas w organizmach
żywych, jest to proces indukowany przez różne czynniki też spontaniczne wynikające ze
niedokładności na przykład procesu replikacji DNA, z transkrypcji też i oczywiście te mutacje
spontaniczne są naprawiane w różnym zakresie ale oczywiście też nie są i to może prowadzić
do zmian nowotworowych i innych problemów. Czyli jedna grupa mutacji są to mutacje
spontaniczne i takie też często są wykorzystywane w laboratorium, do badania w genetyce
szczególnie ale są też takie mutacje, które są indukowane specjalnie w laboratorium. Na
przykład jeżeli jakiś organizm jest naświetlany promieniowaniem wysoko energetycznym
częstość mutacji wzrasta i takie postępowania stosowano i stosuje się bardzo często po to aby
wygenerować mutanty jakieś interesujące. Z kolei te mutacje czy ta mutageneza jest określana
mianem mutagenezy nieukierunkowanej. Natomiast w laboratorium inżynierii genetycznej
inżynierii białka albo (…) molekularnej metody inżynierii genetycznej po prostu są
narzędziem. Najczęściej wprowadzamy mutacje ukierunkowane, zdefiniowane. Technika ta
nosi nazwę (site directed mutagenesis) mutageneza ukierunkowana. Opisowo możemy
powiedzieć ze wprowadzamy mutacje tam gdzie chcemy czyli tak jak to jest na naszej
ilustracji:
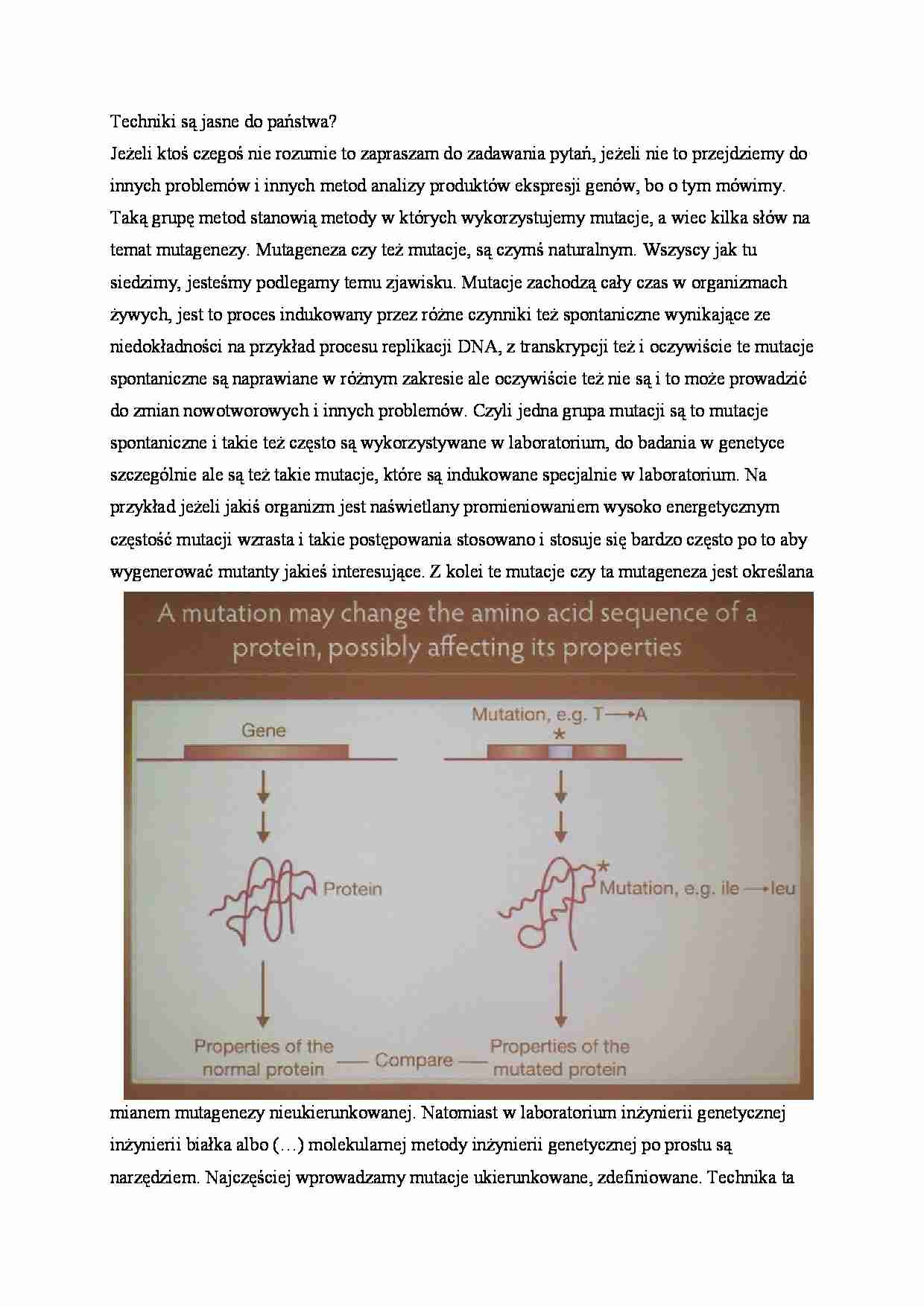
W sekwencji kodującej zamieniamy wybraną resztę i wprowadzamy te zmianę kodonu, istotą
techniki mutagenezy czy też eksperymentu, w którym wykorzystuje się mutagenezę
ukierunkowaną jest po pierwsze wprowadzenie mutacji a po drugie obserwowanie zmiany
jaką ta mutacja wprowadza. Tak jak poprzednio mówiłem to jest możliwe tylko wtedy jeżeli
mamy określony jakiś układ odniesienia, jeżeli możemy porównać do czegoś właściwości tak
zmienionego produktu. Zatem w eksperymencie mutagenezy po pierwsze wprowadzamy
mutację, zamienimy sekwencje kodującą w jakimś tam zakresie, to wprowadza mutację w
białku, na przykładzie izoleucyna została zamieniona na leucynę i otrzymujemy produkt
białkowy o określonych właściwościach. Ale te właściwości są dla nas istotne tylko wtedy
gdy porównamy je z właściwościami białka typu dzikiego. Także każdy eksperyment
mutagenezy polega na, że mamy produkt zmieniony i produkt typu dzikiego i je
porównujemy ze sobą. Tak jak tutaj porównano czy też zilustrowano, te typ dzikiego służą do
wytworzenia białka typu dzikiego określonych właściwościach i te właściwości są ze sobą
zestawiane i porównywane
Teraz chciałem omówić pobieżnie kilka technik mutagenezy ( tutaj wywód że jest to temat
rzeka można by cały semestr wykładać).
Ja po prostu zwrócę uwagę na pewne problemy i metody. Różne są metody wprowadzania
mutacji, na przykładzie który przed chwilą państwu pokazywałem na ilustracji była prosta
mutacja, mutacja punktowa czyli zamiana jednej reszty aminokwasowej na druga, ale mutacje
ukierunkowane to nie tylko zamiany reszt. Mutacje ukierunkowane czyli kierowane przez
badacza mogą prowadzić do delecji fragmentu sekwencji w jakimś białku, do inercji to też
mutacja czyli wbudowujemy w białko sekwencje której tam wcześniej nie było. Może to być
również technika w której jakiś fragment w białku podstawimy fragmentem innego białka to
jest też mutacja, także proszę nie myśleć ze mutacji ukireunkowanej jako tylko o zamianie
jednej reszty. Chodzi o to, że każda z tych metod jest racjonalna, eksperymentator do końca
panuje na tym i wie czego chce. A jeżeli nie panuje to jest to z zamysłem. Wiec pierwszym
przykładem jest mutacja, technika w której zmierzamy do delecji fragmentu genu. Mamy
sytuacje wyjściową czyli układ odniesienia.
Widzą Państwo gen, w którym są jakieś przykładowe dwie sekwencje wyróżnione w tym
przypadku sekwencje Alu i one są tak ułożone w tym genie, że z możemy z nich korzystać do
przeprowadzenia delecji. Czyli wytrawiając enzymem AluI i usuwamy fragment genu
następnie używamy ligazy i otrzymujemy gen z delecją. Oczywiste to wygląda bardzo pięknie
jeżeli taka delecja nie spowoduje przesunięcia ramki odczytu, jeżeli ramka odczytu jest
zachowana to otrzymujemy produkt białkowy który dokładnie odpowiada delecji bez
przesunięcia ramki. To jest pierwszy sposób gdzie korzystamy z miejsca restrykcyjnego aby
otrzymać delecję.
Delecja ale wykonana w trochę inny sposób. Wyobrażamy sobie sytuacje gdzie mamy tylko
jedno miejsce restrycyjne np. EcoR1, nie ma dwóch dowolnych żeby taka sporą delecja jaką
była na poprzednim schemacie można było zrobić. Zatem przy pomocy enzymu
restrykcyjnego przecinamy sekwencje i następnie używamy enzymu przy Bal31 do dokonania
delecji obu produktów. Pamiętacie Państwo, że Bal31 jest taką egzonukleaza, która nie
wykazuje preferencji w odniesieniu do sekwencji i naprawia 3’ i 5’ końce (szczerze mówiąc
ja bym nigdy takiej metody nie użył, ale warto wiedzieć, że jak ktoś bardzo chce to może.
Dlaczego bym nie użył? Dlatego, że nie miałbym kontroli nad tym jak duża jest ta delecja i na
pewno powstałby by tutaj tez produkty, których ramka odczytu byłaby za duża (niewyraźnie?)
ale teoretycznie można otrzymać całą bibliotekę mutantów a potem wybrać te właściwe)
A wiec jeden i drugi produkt EcoR1 są nacinane i traktowane Bal31, potem wprowadzamy
ligazę- one podlegają ligacji i otrzymujemy gen z delecja a w konsekwencji białko z delecją.
Tu jest napisane „protein with a minor deletion” ale to niekoniecznie musi być prawda,
ponieważ to czy ona będzie duża czy mała zależy od czasu w jakim Bal31 działało. Zwrócicie
uwagę, tak zupełnie na marginesie że te „narzędzia”, enzymy którymi się posługujemy są
nam znane, ale od ich układu, kolejności wykorzystania zależy to co… Kolejna metoda
(bardzo ciekawa) otóż tutaj możemy wprowadzić insercję, mianowicie widzimy sekwencję
typu „dzikiego”, gdzie jest znów EcoR1 miejsce.
Przy pomocy EcoR1 trawimy sekwencję i po przecięciu ligujemy z oligonukleotydem , który
oczywiście ma końce komplementarne do sekwencji EcoR1- w ten sposób otrzymujemy gen z
insercją, który daje produkt białkowy już zmieniony. Ten produkt, w tym konkretnym
eksperymencie uległ destabilizacji- wpłynęliśmy jakoś na jego strukturę no i mogło tak być że
się niezestabilizowałą, tutaj są różne powody dla których można taką insercję robić, ale
zostawmy je na boku. Tutaj teraz pokazaliśmy sobie jak można insercję zobaczyć. Z Ilustracji
dodatkowych… ( tu już niewyraźnie, jakieś szmery „ o ja pierdole, rozlało się”)
Metoda mutagenezy zwana metodą mutagenezy kasetowej, otóż jak sama nazwa wskazuje
jest wymieniamy fragment sekwencji kodującej czyli kaseta. Metoda jest niezwykle prosta,
musimy mieć po prostu sekwencję lub gen, któremu chcemy wymienić kasetę i co do tej
sekwencji, na której pracujemy jest tylko jedno wymaganie czyli jedna jakieś założenie, że w
niej występują dwa miejsca restrykcyjne dogodne do tego aby kasetę wymienić. Popatrzcie na
ilustrację.
Pracujemy na tej zielonej sekwencji, umówmy się. Tu było jedno miejsce restrykcyjne a tu
drugie, korzystając z tych z miejsc restrykcyjnych, odtrawiliśmy ten fragment DNA, to jest
nasza kaseta typu dzikiego i na jej miejsce wstawiamy teraz sekwencję zmutowana i to jest ta
sekwencja. Widzimy, że tu jest wprowadzona mutacja, zaznaczona kolorem, po ligacji
dostajemy sekwencję zmutowaną. To byłaby mutageneza kasetowa, jeżeli tylko można tak
postąpić to oczywiście polecałbym dlatego że tutaj efektywność wprowadzenia mutacji jest
niemal 100%. Oczywiście na schemacie mamy bardzo prostu przypadek, wymianie uległa
pojedyncza para zasad, możemy w ogóle wiele par wymienić jednocześnie. Możemy
posługując się taką metodą wprowadzić kasetę zupełnie nieadekwatną dla danego genu
białka. Możemy wprowadzić bardzo długo fragment i wtedy otrzymamy białko nieistniejące
do tej pory.
(niedosłyszane pytanie od publiczności coś o insercjach, odtrawianiu, miejscach
restrykcyjnych- lepiej dwa a nie jedno bo wtedy klonowanie jest ukierunkowane, w tym
czasie ktoś zasnął i spadł z krzesła :D )
Ale proszę Państwa ta metoda niepozorna ze schematów ma przeogromny potencjał,
mianowicie, wyobraźcie sobie Państwo eksperyment następujący: trawimy, usuwamy tą
sekwencję typu dzikiego a na jej miejsce wstawiamy niejeden fragment o określonej
sekwencji, nie używamy do ligacji jednego fragmentu o określonej ale całą biblioteką, a więc
zrandomizowane sekwencje czyli pracujemy z całą biblioteką. Na przykład wszystkich
przypadkowych sekwencji, które dotyczą jakiegoś fragmentu kodującego fragment białka.
Wyobraźmy sobie problem, bardzo praktyczny, mamy gen na przykład jakiejś lipazy, która
jest lipazą którą możemy potencjalnie wykorzystać w proszku, który zawiera ( jak to się
pięknie mówi w reklamach) nową formułę (nikt nie wie co to jest ta formuła, może chodzi o
jakiś nowy związek chemiczny albo właśnie nowy enzym) ale trudno oczekiwać, aby Pan czy
tez Pani z reklamy tłumaczyła, że właśnie otrzymaliśmy nową wersję enzymu lipazy
metodami inżynierii genetycznej i dlatego ten proszek będzie w jeszcze niższej temperaturze,
jeszcze szybciej usuwał te wstrętne plamy z ukochanej koszulki dziecka, albo mamy
(koszulkę nie mamę mam nadzieję?) którą dziecko użyło do wytarcia podłogi. Mamy problem
z tym żeby wprowadzić ten proszek z nową formułą na rynek i poszukujemy nowej lipazy i
poznaliśmy gen, poznaliśmy tę sekwencję, która jest istotna dla centrum katalitycznego
pamiętając ciągle że właśnie mamy jakiś deadline (…) Chcemy ulepszyć enzym, ale
ulepszanie enzymów rzadko jest procesem racjonalnym, oczywiście są takie opowieści o
racjonalnych ulepszeniach, ale metody racjonale ulepszania enzymów nie są pewne zatem
bardzo często stosuje się metody randomiczne czyli własnie takie że korzysta się z metod
mutagenezy w której jednocześnie generujemy cała szereg mutantów a więc jeżeli byśmy
mieli gen, w którym akurat jakis fragment odpowiada za kodowanie reszty istotnie dla
kataliazy tego wspomnianego enzymu.
... zobacz całą notatkę

Komentarze użytkowników (0)