To tylko jedna z 2 stron tej notatki. Zaloguj się aby zobaczyć ten dokument.
Zobacz
całą notatkę

Wyciszanie genów u grzybów strzępkowych
Późno rozwinięty dział
Rozwój dopiero po odkryciu zjawiska interferencji RNA - iRNA
1. RIP
Proces typowy dla grzybów
proces oparty na homologii, który powoduje mutację powtarzającego się DNA często prowadzi do epigenetycznego wyciszana zmutowanych sekwencji przez metyzację DNA.
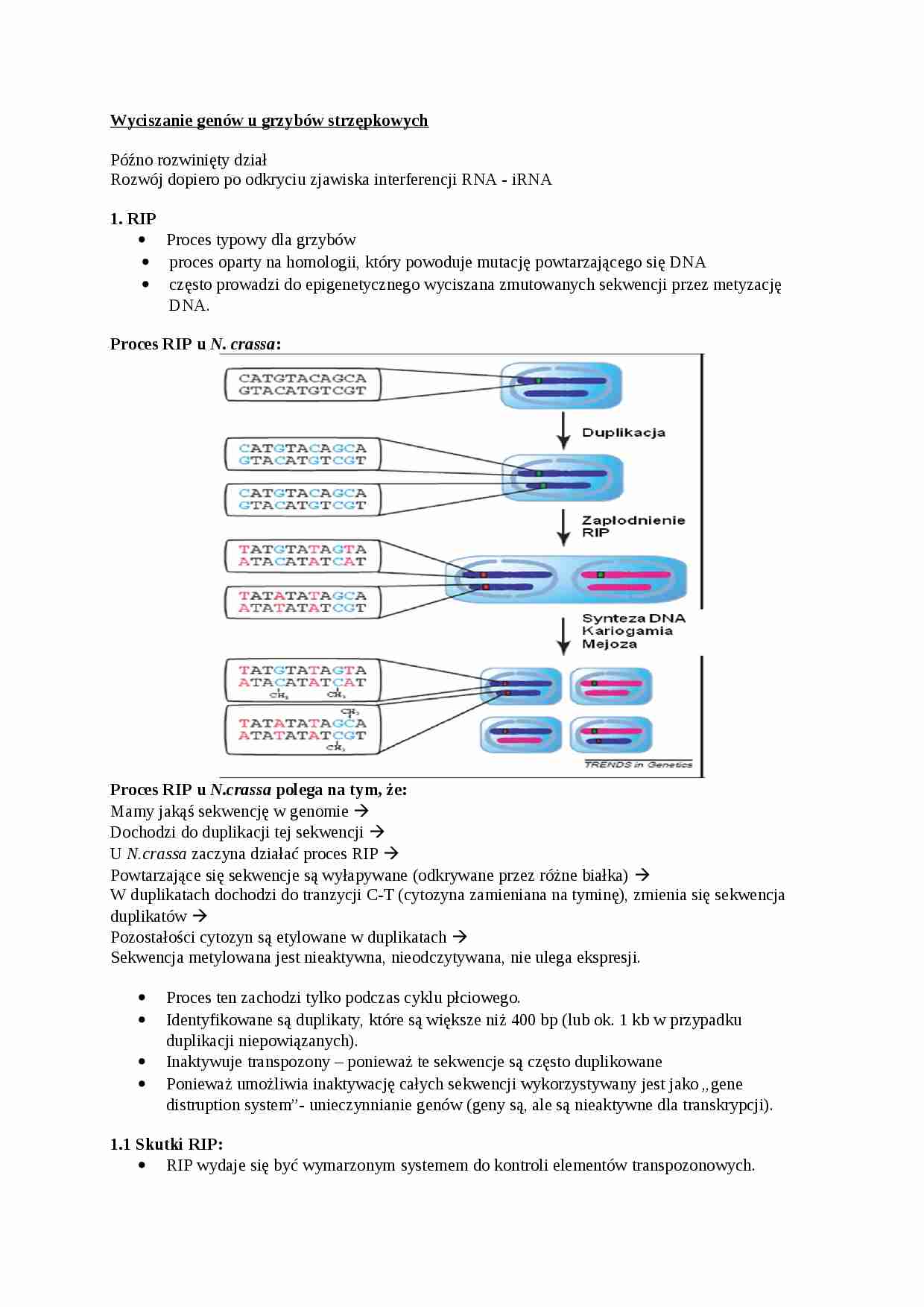
Proces RIP u N. crassa:
Proces RIP u N.crassa polega na tym, że:
Mamy jakąś sekwencję w genomie Dochodzi do duplikacji tej sekwencji U N.crassa zaczyna działać proces RIP Powtarzające się sekwencje są wyłapywane (odkrywane przez różne białka) W duplikatach dochodzi do tranzycji C-T (cytozyna zamieniana na tyminę), zmienia się sekwencja duplikatów Pozostałości cytozyn są etylowane w duplikatach Sekwencja metylowana jest nieaktywna, nieodczytywana, nie ulega ekspresji.
Proces ten zachodzi tylko podczas cyklu płciowego.
Identyfikowane są duplikaty, które są większe niż 400 bp (lub ok. 1 kb w przypadku duplikacji niepowiązanych).
Inaktywuje transpozony - ponieważ te sekwencje są często duplikowane
Ponieważ umożliwia inaktywację całych sekwencji wykorzystywany jest jako „gene distruption system”- unieczynnianie genów (geny są, ale są nieaktywne dla transkrypcji).
1.1 Skutki RIP:
RIP wydaje się być wymarzonym systemem do kontroli elementów transpozonowych.
Z drugiej str RIP wydaje się przeszkadzać w ewolucji genomu, biorąc pod uwagę fakt, że duplikacje genów są podstawą do dostarczenia materiału do ewolucji (przystosowywanie się do nowych warunków środowiska przez mutacje).
1.2 Czy elementy zalegają w genomie?:
Wykazano, że u N.crassa istnieje niewielka ilość par genów o dużej homologii.
Nie ma podobnych genów
Im większe podobieństwo sekwencji, tym system RIP mniej atakuje. Jak obserwujemy podobieństwo w 40% to RIP już wcale nie atakuje takiej sekwencji - jest to cecha charakterystyczna dla N. crassa. Spośród 10 tys genów kodujących białka N.crassa, tylko 6 par (12 genów) ma homologiczne 80% nukleotydów.
Niesprzężoną parę genów histonu H4
Niesprzężoną parę genów białka rybosomalnego L40e
3 pary domniemanych białek, kodujących krótsze sekwencje niż oczywiste minimum wymagane przez RIP
Ostatnia para wysoce podobnych genów to para syntez glutaminowych, które mają zupełnie inny układ exonów i intronów i w związku z tym nigdy nie dzielą regionów genomu „wrażliwych” na RIP (np. 400 bp z 80% identycznością nukleotydów)
(…)
…, wyniki te są zgodne z obserwowanym brakiem transpozycji w licznych dzikich szczepach Neurospora
Analiza sekwencji regionów centromerowych grupy sprzężenia VII, wykazała obecność reliktów kilku dodatkowych typów transpozonów, a badanie genomowych sekwencji związanych z metylacją DNA ujawniło występowanie jeszcze większego zestawu transpozonów zinatywowanych przez RIP
Retrotranspozony wymagają odwrotnej…
... zobacz całą notatkę




Komentarze użytkowników (0)