

Fragment notatki:
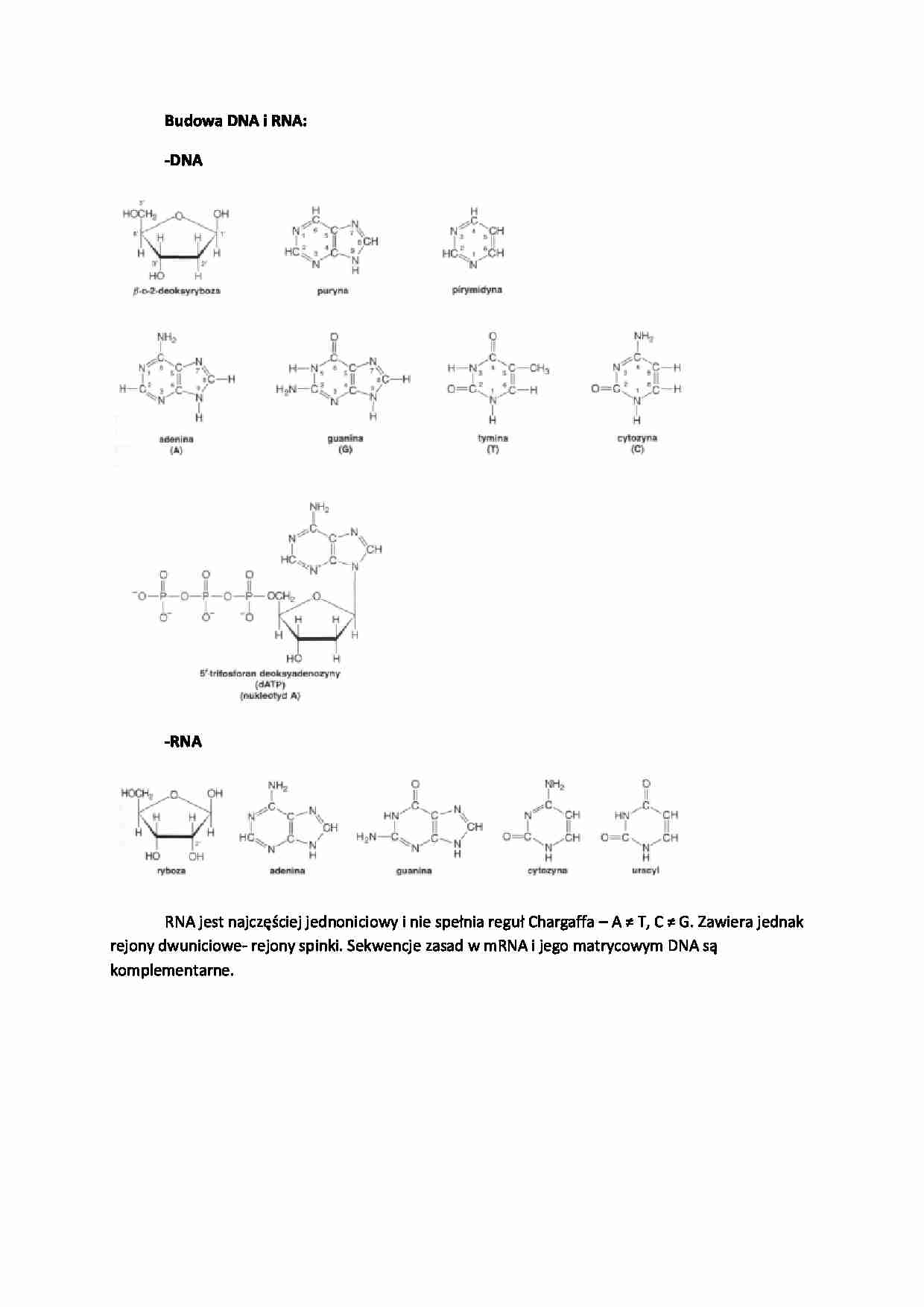
Budowa DNA i RNA:
-DNA
-RNA
RNA jest najczęściej jednoniciowy i nie spełnia reguł Chargaffa – A ≠ T, C ≠ G. Zawiera jednak
rejony dwuniciowe- rejony spinki. Sekwencje zasad w mRNA i jego matrycowym DNA są
komplementarne.
Budowa helisy DNA-porównanie
BIAŁKA UCZESTNICZĄCE W KLONOWANIU, SYNTEZIE, REPLIKACJI I NAPRAWIE DNA
Polimeraza DNA I wymaga:
- 4 dNTP
- Mg2+
-matrycy
- primera z wolną grupą 3’-OH
Polimeryzacja następuje w kierunku 5’→ 3’ poprzez atak grupy 3’-OH na grupę alfa
fosforanową nukleotydu- tworzy się wiązanie fosfodiestrowe.
Polimeraza I posiada aktywnośd nukleazową w kierunkach:
- 5’→ 3’ dzięki, której usuwany jest odcinek starterowy RNA, usuwanie 12 nukleotydowego
fragmentu w celu wycięcia dimerów pirymidynowych (naprawa DNA)
- 3’→5’ umożliwiającą korektę błędów syntezy (polimeraza sprawdza wynik każdego
przyłączenia nukleotydu przed przystąpieniem do następnego cyklu).
Polimerazy II i III wymagają:
- matrycy
- 4 dNTP
- primera z wolną grupą 3’-OH
- Mg 2+
Wielopodjednostkowy kompleks zawierający polimerazę III syntezuje większośd nowego DNA
, natomiast polimeraza I usuwa startery i uzupełnia przerwy, polimeraza II współdziała podczas
naprawy DNA ale nie jest potrzebna do replikacji.
Holoenzym polimerazy III katalizuje tworzenie tysięcy wiązao fosfodiestrowych bez
oddysocjowania od matrycy, jest on asymetrycznym dimerem ponieważ obie nici – prowadząca i
opóźniona – muszą ulegad replikacji w tym samym czasie. Podjednostka alfa jest polimerazą
natomiast podjednostka epsilon jest egzonukleazą 3’→5’ sprawdzającą poprawnośd replikacji. Aby
obie nici były syntezowane w kierunku 5’→3’ nid opóźniona musi się utworzyd pętlę wokół jednej z
podjednostek holoenzymu.
Odwrotna transkryptaza - polimeraza DNA zależna od RNA. RNA jest matrycą dla DNA (-)
następnie dosyntezowane DNA jest matrycą dla kolejnej nici DNA (+) (3 aktywności polimeraza DNA
zależna od RNA, polimeraza DNA zależna od DNA i RNAza). Odwrotna transkryptaza wymaga:
-primera sparowany z RNA z wolną grupą 3’-OH
-4 DNTP
Polmeraza RNA zależna od DNA wymaga:
- 4 NTP
- Mg 2+
- matrycy dwuniciowy DNA ( ssRNA, dsRNA,hybryd RNA-DNA nie jest matrycą‼!)
Polimeryzacja następuje w kierunku 5’→ 3’ poprzez atak grupy 3’-OH na grupę alfa
fosforanową nukleotydu- tworzy się wiązanie fosfodiestrowe. Nie jest wymagany primer. Synteza
RNA jest w pełni konserwatywna.
Endonukleazy restrykcyjne rozpoznają specyficzne sekwencje zasad ( najczęściej
palindromowe -posiadające podwójną symetrię obrotową) w dwuniciowej helisie i zrywają obie nici
w dokładnie zdefiniowanych miejscach. Rozcięcie może posiadad kooce tępe lub lepkie-kohezyjne.
Endonukleazy przeszukując duży rowek DNA szybko i bardzo precyzyjnie odczytują sekwencje
nukleotydową.
Ligazy DNA katalizują tworzenie wiązao fosfodiestrowych w miejscach zerwania łaocucha
DNA.Ligazy wymagają:
- wolnej grupy 3’-OH
- ufosforylowanej grupy 5’-OH
- ATP/ NAD+
Ligaza tworzy wiązanie poprzez związanie ATP/NAD+ do Lys ,tworzy się produkt pośredni
AMP-enzym, następnie ta aktywowana grupa jest przenoszona na koniec 5’ łaocucha DNA,tworzy się
5’-P-AMP, i dalej ligaza katalizuje tworzenie wiązania fosfodiestrowego poprzez atak grupy 3’-OH na
aktywowany koniec 5’-P
Replikacja DNA jest semikonserwatywna tzn. że jedna nid jest rodzicielska, a druga potomna.
U procaryota synteza DNA zachodzi w obu kierunkach od rejonu inicjacji -oriC- bogatego w
pary AT. Replikacja zaczyna się od przyłączenia białka dnaA do rejonów bogatych a AT. Białka dna B i
C łączą się z białkiem dnaA aby wygiąd i otworzyd dwuniciową helisę. Białko dnaB jest helikazą, która
rozplata helisę kosztem ATP. Następnie białko SSB wiąże się do rozplecionej nici zapobiegając
skręceniu się ich z powrotem. Rozwijanie kolistego DNA na początku procesu powoduje tworzenie
dodatnich superskrętów, które są usuwane przez gyrazę (hydroliza ATP).
Aby rozpocząd syntezę DNA na obu niciach potrzebny jest starter, którym jest krótki odcinek
RNA tworzony przez prymazę (która po wytworzeniu primera przesuwana jest po nici opóźnionej w
kierunku 5’→3’ wytwarzając kolejne startery do syntezy fragmentów Okazaki). Nid 5’→3’ jest
dobudowyana ciągle i jest nazywana nicią prowadzącą, natomiast nid 3’→5’ określana jest jako nid
opóźniona ze względu na na fakt, że jej znaczna częśd występuje w postaci krótkich fragmentów
(Okazaki) następnie łączonych przez ligazę. Nieciągła synteza nici opóźnionej pozwala na
polimeryzację w kierunku 5’→3’ na poziomie nukleotydowym, co daje ogólny kierunek wydłużania
nici 3’→5’.
U Prokaryota transkrypcja rozpoczyna się od promotora TATAAT położonych 10 par zasad
przed samym genem lub sekwencji TTGACA znajdującej się 35 par zasad przed genem ulegającym
transkrypcji. U Eucaryota podobna kaseta TATA znajduje się ok. 25 par zasad przed genem oraz
kaseta CAAT ok. 75 par zasad przed transkrybowanym genem dodatkowo u Eucaryota znajdują się
sekwencje wzmagające. Zakooczenie transkrypcji w miejscu terminacji, w którym nowo
zsyntezowana nid RNA przybiera postad spinki i spontanicznie oddysocjowuje od enzymu.
Kod genetyczny jest:
- trójkowy -64 trójki, 61 oznacza aminokwasy, 3 oznaczają koniec łaocucha UAA UAG i UGA,
tylko tryptofan i metionina ( AUG też sygnał start) kodowane są przez 1 trójkę;
-bezprzecinkowy;
-nienakładający się;
-zdegenerowany;
Kodony kodujące tan sam aminokwas nazywamy synonimami. Najczęściej synonimy różnią
się ostatnią zasadą trypletu.
Sekwencja zasad w genie i sekwencja aminokwasów w jego białku są wspóliniowe.
BADANIA DNA
Aby zidentyfikowad określony fragment DNA można go pociąd różnymi endonukleazami,
rozdzielid wg. wielkości za pomocą elektroforezy, zrobid odbitkę na arkuszu nitrucelulozowym,
następnie dodad sondę znakowaną P32 komplementarną do interesującego nas fragmentu DNA i
wykonad autoradiografię (Southen blotting, Northern Blotting – oznaczanie RNA; Western blottingoznaczanie białek)
Metoda Sangera- metoda sekwencjonowania DNA- wymaga:
- matrycy DNA (sekwencjonowany DNA)
- polimerazy I DNA
- 4 dNTP
- primera
- 4 ddNTP
Wbudowanie ddNTP do rosnącego łaocucha uniemożliwa jego dalsze wydłużanie, ponieważ
analog ten nie zawiera grupy 3’-OH. Poprzez odpowiednie dobranie stężeo reagentów powstają
fragmenty DNA o różnej długości, w których ddNTP znajduje się na koocu 3’. Mieszaninę następnie
poddaje się elektroforezie i odczytuje sekwencję badanego DNA (zsenkwenjonowany fragment jest
komplementarny do badanej nici) na autoradiogramie.
Rekombinacja DNA za pomocą wektorów. Do klonowania DNA wybiera się głównie plazmidy
lub faga λ. Odpowiedni fragment wektora zostaje przecięty przez enzym restrykcyjny, do takiego
zmodyfikowanego plazmidu można wprowadzid dowolny fragment DNA pod warunkiem, że ma takie
same „kooce” jak rozcięty wektor.
Inaktywacja insercyjna – inaktywowanie genów np. oporności w plazmidzie poprzez
przecięcie go endonukleazą. (Selekcja bakterii)
Po rozdzieleniu genomowego DNA na łaocuchy o wielkości mniej więcej 15 kz można je
włączyd w genom wirusa i utworzyd populację zrekombinowanych fagów zawierających prawie
kompletny genom – biblioteka genomowa. Wyszukiwanie interesujących nas genów poprzez
odwzorowanie układu łysinek na nitrocelulozie poddanej lizie i zastosowaniu znakowanych sond
DNA.
Biblioteka cDNA- biblioteka genów zawierająca tylko DNA ekspresjonowane w danej
komórce. Tworzy się ją poprzez izolowanie całego mRNA z komórki, przekształceniu do DNA przez
odwrotną transkryptazę, a następnie klonowanie w wektorze np. wirusie. Aby uzyskad maksymalną
transkrypcję, cDNA wprowadza się do wektora blisko silnego promotora bakteryjnego. Obecnośd
klonów DNA można ujawnid na podstawie ich zdolności do kierowania syntezą białka obcego dla
bakterii będącej gospodarzem (wykrywanie przez zastosowanie autoradiogramu z repliki kolonii ze
znakowanymi przeciwciałami swoistymi dla białek)
PCR – reakcja łaocuchowej polimeryzacji, służy do wielokrotnego powielenia (ampiflikacji)
sekwencji DNA . PCR wymaga:
- DNA
- 4 dNTP
- 2 primerów, których sekwencja otacza fragment DNA nas interesujący
- termostabilnej polimerazy DNA
Metoda PCR polega na wielokrotnym powtarzaniu poniższych kroków co prowadzi do
ogromnego zwielokrotnienia interesującej nas (znajdującej się miedzy primerami) sekwencji DNA:
- ogrzanie do temperatury rozdzielenia nici DNA
- szybkie ochłodzenie mieszaniny, dodanie do niej sekwencji primerowych co skutkuje
dołączeniem ich do komplementarnych rejonów DNA
- synteza komplementarnego DNA przez polimerazę DNA
Wszystkie nowe cząsteczki DNA służą jako matryce w kolejnych cyklach. Jedynym DNA, który
ulega amplifikacji jest odcinek zawarty pomiędzy parą primerów zhybrydyzowanych z wyjściowym
DNA.
Ukierunkowana mutageneza. Po izolacji genu możliwe jest zmodyfikowanie sekwencji w celu
zmiany zakodowanej sekwencji aminokwasów. Aby skorzystad z tej techniki potrzebujemy
oligonukleotydu, którego sekwencja jest komplementarna do interesującego nas fragmentu genomu
jednak posiada zmienioną zasadę azotową. Zhybrydyzowany do rozdzielonego plazmidu
oligonukleotyd służy jako primer dla polimerazy DNA, a produkt syntezy zostaje zamknięty jako
cząsteczka kolista (przez ligazę) i służy jako zmutowany plazmid.
Mutacje:
- Substytucja – podstawienie jednej pary zasad zamiast innej, rodzaje:
-tranzycja – podstawianie jednej puryny w miejsce drugiej lub pirymidyny w miejsce
drugiej pirymidyny A→G, G→A, C→T, T→C
- transwersja – zamiana pirymidyny na purynę lub puryny na pirymidynę
- Delecja – usunięcie jednej lub większej ilości par zasad
- Insercja – wstawienie jednej lub większej ilości par zasad.
Mutageny :
- 5-bromouracyl – powstawanie tranzycji AT→GC lub GC→ AT
- 2-aminopuryna – powstawanie tranzycji AT→GC lub GC→ AT
- kwas azotowy (III) – powstawanie tranzycji AT→GC lub GC→ AT
- hydroksyloamina – jednokierunkowa tranzycja GC→ AT
- akrydyny – delecje lub insercje jednej lub więcej par nukleotydów
- UV – powstawanie dimerów pirymidynowych CC, TT
... zobacz całą notatkę

Komentarze użytkowników (0)