

Fragment notatki:
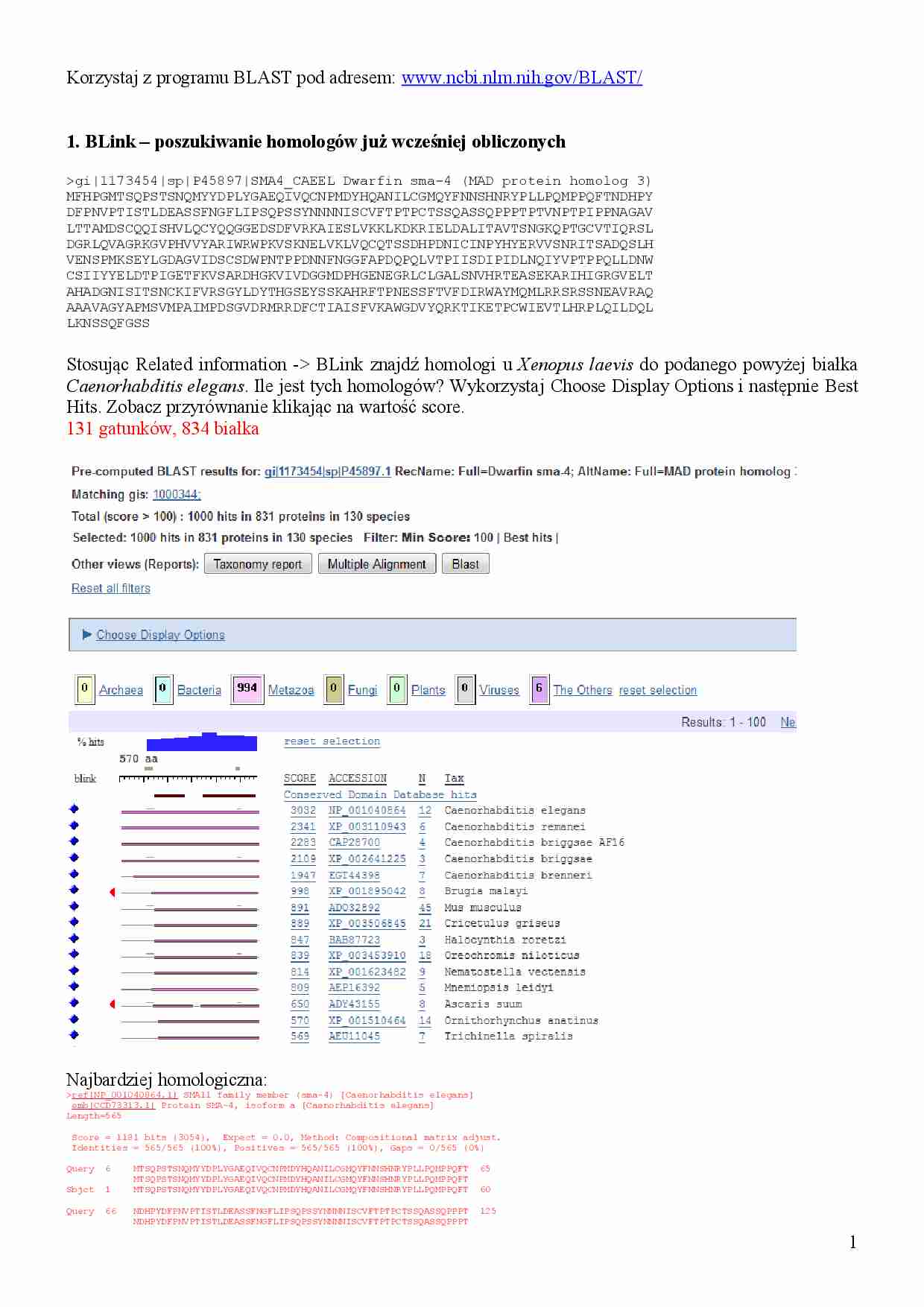
Korzystaj z programu BLAST pod adresem: www.ncbi.nlm.nih.gov/BLAST/ 1. BLink – poszukiwanie homologów już wcześniej obliczonych gi|1173454|sp|P45897|SMA4_CAEEL Dwarfin sma-4 (MAD protein homolog 3) MFHPGMTSQPSTSNQMYYDPLYGAEQIVQCNPMDYHQANILCGMQYFNNSHNRYPLLPQMPPQFTNDHPY DFPNVPTISTLDEASSFNGFLIPSQPSSYNNNNISCVFTPTPCTSSQASSQPPPTPTVNPTPIPPNAGAV LTTAMDSCQQISHVLQCYQQGGEDSDFVRKAIESLVKKLKDKRIELDALITAVTSNGKQPTGCVTIQRSL DGRLQVAGRKGVPHVVYARIWRWPKVSKNELVKLVQCQTSSDHPDNICINPYHYERVVSNRITSADQSLH VENSPMKSEYLGDAGVIDSCSDWPNTPPDNNFNGGFAPDQPQLVTPIISDIPIDLNQIYVPTPPQLLDNW CSIIYYELDTPIGETFKVSARDHGKVIVDGGMDPHGENEGRLCLGALSNVHRTEASEKARIHIGRGVELT AHADGNISITSNCKIFVRSGYLDYTHGSEYSSKAHRFTPNESSFTVFDIRWAYMQMLRRSRSSNEAVRAQ AAAVAGYAPMSVMPAIMPDSGVDRMRRDFCTIAISFVKAWGDVYQRKTIKETPCWIEVTLHRPLQILDQL LKNSSQFGSS Stosując Related information - BLink znajdź homologi u Xenopus laevis do podanego powyżej białka Caenorhabditis elegans . Ile jest tych homologów? Wykorzystaj Choose Display Options i następnie Best Hits. Zobacz przyrównanie klikając na wartość score. 131 gatunków, 834 białka Najbardziej homologiczna: ref|NP_001040864.1| SMAll family member (sma-4) [Caenorhabditis elegans] emb|CCD73313.1| Protein SMA-4, isoform a [Caenorhabditis elegans] Length=565 Score = 1181 bits (3054), Expect = 0.0, Method: Compositional matrix adjust. Identities = 565/565 (100%), Positives = 565/565 (100%), Gaps = 0/565 (0%) Query 6 MTSQPSTSNQMYYDPLYGAEQIVQCNPMDYHQANILCGMQYFNNSHNRYPLLPQMPPQFT 65 MTSQPSTSNQMYYDPLYGAEQIVQCNPMDYHQANILCGMQYFNNSHNRYPLLPQMPPQFT Sbjct 1 MTSQPSTSNQMYYDPLYGAEQIVQCNPMDYHQANILCGMQYFNNSHNRYPLLPQMPPQFT 60 Query 66 NDHPYDFPNVPTISTLDEASSFNGFLIPSQPSSYNNNNISCVFTPTPCTSSQASSQPPPT 125 NDHPYDFPNVPTISTLDEASSFNGFLIPSQPSSYNNNNISCVFTPTPCTSSQASSQPPPT 1 Sbjct 61 NDHPYDFPNVPTISTLDEASSFNGFLIPSQPSSYNNNNISCVFTPTPCTSSQASSQPPPT 120 Query 126 PTVNPTPIPPNAGAVLTTAMDSCQQISHVLQCYQQGGEDSDFVRKAIESLVKKLKDKRIE 185 PTVNPTPIPPNAGAVLTTAMDSCQQISHVLQCYQQGGEDSDFVRKAIESLVKKLKDKRIE Sbjct 121 PTVNPTPIPPNAGAVLTTAMDSCQQISHVLQCYQQGGEDSDFVRKAIESLVKKLKDKRIE 180 Query 186 LDALITAVTSNGKQPTGCVTIQRSLDGRLQVAGRKGVPHVVYARIWRWPKVSKNELVKLV 245 LDALITAVTSNGKQPTGCVTIQRSLDGRLQVAGRKGVPHVVYARIWRWPKVSKNELVKLV Sbjct 181 LDALITAVTSNGKQPTGCVTIQRSLDGRLQVAGRKGVPHVVYARIWRWPKVSKNELVKLV 240 Query 246 QCQTSSDHPDNICINPYHYERVVSNRITSADQSLHVENSPMKSEYLGDAGVIDSCSDWPN 305 QCQTSSDHPDNICINPYHYERVVSNRITSADQSLHVENSPMKSEYLGDAGVIDSCSDWPN Sbjct 241 QCQTSSDHPDNICINPYHYERVVSNRITSADQSLHVENSPMKSEYLGDAGVIDSCSDWPN 300 Query 306 TPPDNNFNGGFAPDQPQLVTPIISDIPIDLNQIYVPTPPQLLDNWCSIIYYELDTPIGET 365 TPPDNNFNGGFAPDQPQLVTPIISDIPIDLNQIYVPTPPQLLDNWCSIIYYELDTPIGET Sbjct 301 TPPDNNFNGGFAPDQPQLVTPIISDIPIDLNQIYVPTPPQLLDNWCSIIYYELDTPIGET 360 Query 366 FKVSARDHGKVIVDGGMDPHGENEGRLCLGALSNVHRTEASEKARIHIGRGVELTAHADG 425
(…)
…
GCGTTAGGCGGAAGAGCATATACAATTTCGACATAAATATTTTTTAAACATTTTACATTTTACATTGGTG
TGAAAAGTTTGTACGCTCCAGACATACACATATGCATATATCTAAACACATTCCCGATTTCGAAAATATT
TTAGAACTTTTAGCTTACATTCTTTGTTCTGTTTTTTTGATTTAACCCGTGTAATTATTAATTAATGAAT
AAACTTAAATGCGAAAATGTTCAA
Przeprowadź przeszukiwania bazy nr ssaków w celu znalezienia homologów do genu muszki owocowej
stosując programy MegaBLAST i discontiguous MegaBLAST. Czy są różnice w poszukiwaniach? Z
czego one wynikają?
MegaBLAST – tylko 1 wynik:
discontinuous MegaBLAST:
10
Discontigous MegaBLAST znalazł więcej homologów, ponieważ pozwala na zmianę w co trzecim
nukleotydzie, gdyż nie zmienia to aminokwasu, to zwieksza czułość poszukiwań.
8. Startery do PCR
>F12
GTCAAGTGGCAACTCCGTCAG
>R8
TTGAGAGATGGATTGTTGCTC
11.1. Sprawdź, jaki gen będą amplifikować podane startery. Czy będą namnażać tylko jeden gen? Nie, 2.
Czy są specyficzne? Tak MegaBLAST…
… do podanej sekwencji uwzględniając i
nieuwzględniając filtr na małą złożoność sekwencji. Jakie widzisz różnice?
Bez filtra:
Z filtrem:
6
Filtr odrzucił regiony o małej złożoności (m. in. powtarzające się pojedyncze aminokwasy), które
mogłyby fałszywie wskazać homologi.
4. Wykorzystanie opcji CDS feature
>gi|30260195:120962-121270 Bacillus anthracis str. Ames; 30S ribosomal protein S10…
… przycisku BLAST wejdź do Formatting options i
zaznacz opcję CDS feature. Policz ile jest substytucji synonimicznych a ile niesynonimicznych w
pierwszym najdłuższym przyrównaniu.
40 synonimicznych
6 niesynonimicznych
5. Przeszukiwanie baz wybranych organizmów przez BLASTP
>gi|20302034|ref|NP_620227.1| MAD homolog 9 [Rattus norvegicus]
MHPSTPISSLFSFTSPAVKRLLGWKQGDEEEKWAEKAVDSLVKKLKKKKGAMDELERALSCPGQPSKCVT…
… do Map Viewer. Jeśli przy ustawieniach domyślnych się nic nie
znajdzie zmień parametry E-value (Expect) na odpowiednią wartość i wybierz opcję BLASTN. Dlaczego
trzeba zmienić parametry? Sekwencja jest zbyt krótka, aby znalazł ją algorytm MegaBLAST. Na jakim
chromosomie i w jakich częściach pewnego genu człowieka znajdują się startery? Na 3, gen tumor
necrosis factor ligand superfamily member 1. Jakiej…
… zakodowane jest pewne zdanie w postaci insercji. Zakodowane jest ono
w sekwencji aminokwasowej. Żeby to odczytać wykorzystaj odpowiedni program (BLASTX),
przeszukując podaną sekwencją bazę białkową.
14
SEKRETY SKRYTE SA W SEKWENCIACH DNA
11. Poszukiwanie zanieczyszczeń w sekwencjach
Podczas klonowania i sekwencjonowania może dojść do zanieczyszczeń wektorami, plazmidami lub
innymi sekwencjami…
... zobacz całą notatkę

Komentarze użytkowników (0)