To tylko jedna z 44 stron tej notatki. Zaloguj się aby zobaczyć ten dokument.
Zobacz
całą notatkę


Modyfikacje enzymów
Wykład 4
Produkcja rekombinowanych białek
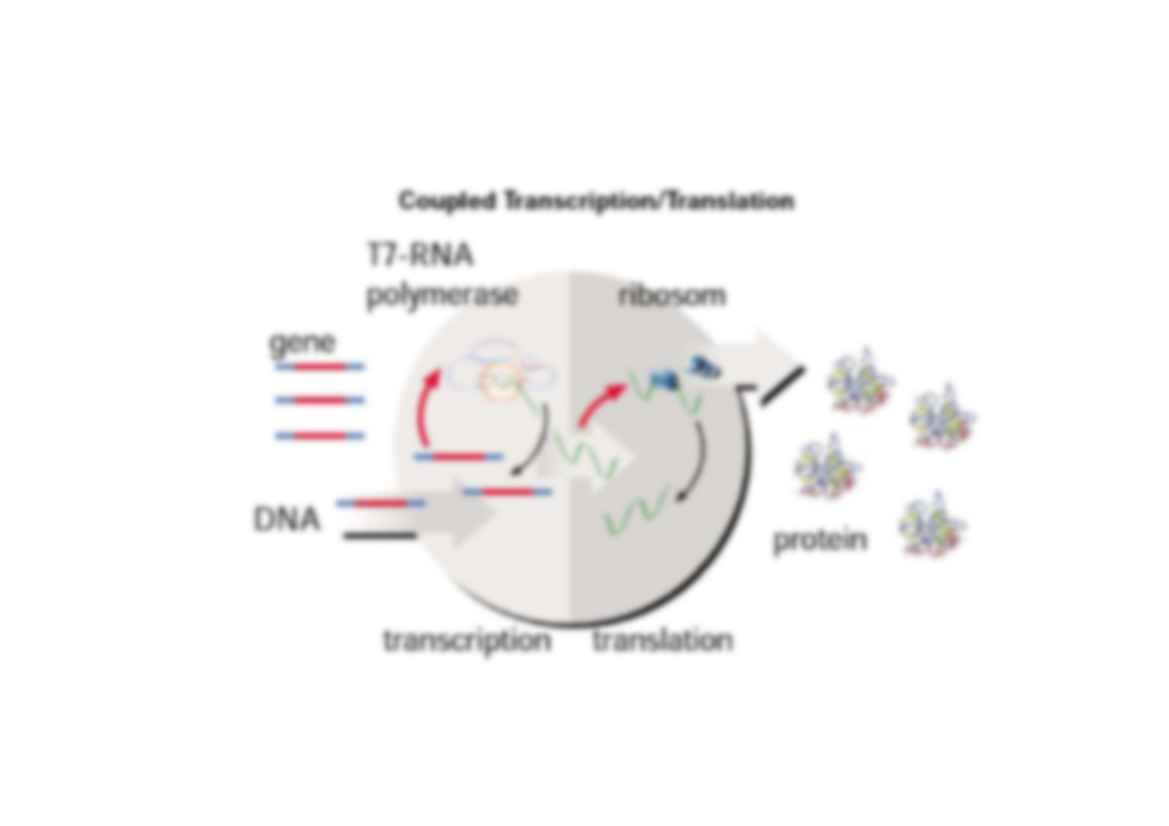
Cell free expression systems
Systemy bezkomórkowe oparte na ekstrakcie z E.coli zostały po raz
pierwszy zastosowane w roku 1973. Przez długi czas były zapomniane z
uwagi na bardzo szybką degradację mRNA.
Obecnie stosuje się systemy sprzężonej transkrypcji-translacji, które
zawierają:
Frakcję rybosomową 30S wraz z niezbędnymi czynnikami do translacji
Polimerazę mRNA faga T7 lub SP6
Jako matrycowe DNA mogą być stosowane plazmidy lub produkty PCR
Wydajność: 20µg białka z 50µl reakcji lub 500-6000µg białka z 1ml
reakcji
Na takiej samej zasadzie stworzono sprzężone systemy eukariotyczne:
Lizat z retikulocytów królika
Ekstrakt z kiełków pszenicy
Cell free expression systems
Cell free expression systems
Wydajna ekspresja białek o wielkościach 10-120 kDa
System umożliwia koekspresję kilku białek jednocześnie z plazmidów z tej
samej grupy kompatybilności
Wygodny system do syntezy białek toksycznych lub trudnorozpuszczalnych
Skrócenie czasu ekspresji do kilku godzin
HIF1α
CK2α
CK2β
HIF1α-pETDuet1
VHL-pGEX4T3,
CK2α-pT7-7,
CK2α’-pETDuet1,
CK2β-pT7-7
CK2α’
Regulacja translacji
Inicjacja translacji: kodony start AUG - 83% genów; GUG – 14%; UUG
– 3% Efektywność: 2-3 razy większa dla AUG, dla GUG niewiele
większa niż dla UUG
Sekwencja Shine-Dalgarno – optymalna inicjacja ekspresji dla
sekwencji 5’-UAAGGAGG-3’ położonej 7±2nt powyżej kodonu AUG
Zasadnicze znaczenie dla efektywności translacji na odległość między
sekwencją SD a kodonem inicjującym start, powinna wynosić 4-14nt
Regulacja translacji
Jeżeli sekwencja SD wchodzi w skład struktur drugorzędowych wydajność
spada – rejon ten powinien być bogaty w reszty A i T
Efektywność translacji można wzmocnić – enhancery - kilkunukleotydwe
sekwencje niektórych bakteriofagów działają jako bardzo efektywne
sekwencje SD w E.coli (wzrost efektywności ekspresji 40-340 razy);
podobny efekt można uzyskać umieszczając taką sekwencję powyżej
naturalnej SD (110-krotny wzrost) lub poniżej (+1 do +6) kodonu AUG
(sekwencja DB (donwstream box)) – sekwencje komplementarne do zasad
16S rRNA.
Bogaty w uracyl 5’-UTR powyżej sekwencji SD może działać także jako
enhancer
Regulacja translacji - stabilność mRNA
Proces degradacji mRNA jest głównym punktem kontroli ekspresji genów.
Okres półtrwania mRNA w komórkach E.coli wynosi od kilku sekund do
20min – nie ma zależności pomiędzy długością cząsteczki a czasem życia.
mRNA degradowany jest przez RNazy - RNaza II i PNPaza (egzonukleazy
3’ 5’) i endonukleazę RNazaE. Nie zidentyfikowano egzonukleazy 5’ 3’.
Enzymy te tworzą kompleks nazywany degradosomem
Regulacja translacji - stabilność mRNA
Można zastosować szczepy posiadające mutacje w genach kodujących
Rnazy, jednakże nie zostało jednoznacznie udowodnione, że brak tych
enzymów wpływa na pulę mRNA
Sekwencja 5’-UTR tworząca struktury spinki do włosów teoretycznie
zabezpiecza przed endonukleazami, ale może osłabić efektywność
translacji - sekwencje stabilizujące
... zobacz całą notatkę






Komentarze użytkowników (0)