To tylko jedna z 9 stron tej notatki. Zaloguj się aby zobaczyć ten dokument.
Zobacz
całą notatkę


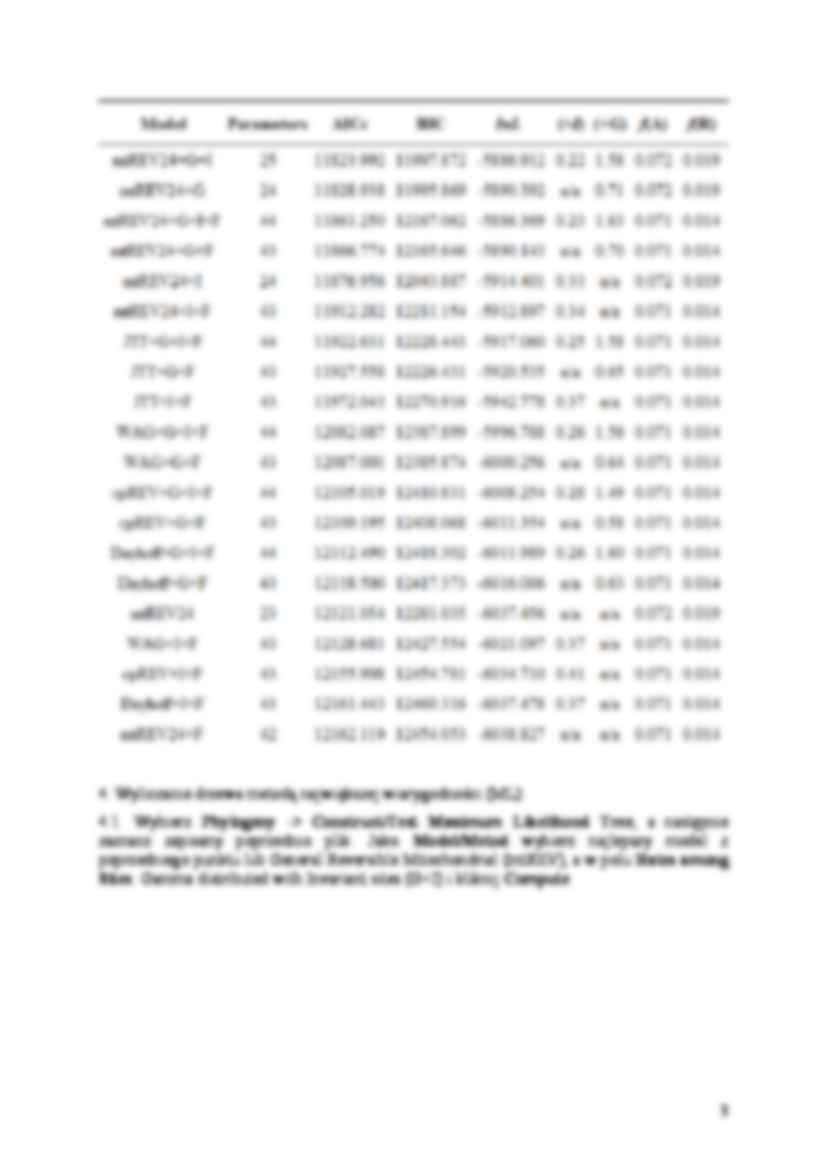
Problemy do rozwiązania 1. Pochodzenie człowieka W oparciu o podaną sekwencję białka mitochondrialnego określ z jakimi małpami człekokształtnymi człowiek jest najbardziej spokrewniony. Przy wyborze sekwencji homologicznych wybierz sekwencje należące do naczelnych (człowieka, szympansa, goryla, orangutana, gibbona, małp zwierzokształtnych), ssaków łożyskowych, torbaczy i stekowców (grupę zewnętrzną). gi|17981863|ref|NP_536853.1| NADH dehydrogenase subunit 5 [Homo sapiens] MTMHTTMTTLTLTSLIPPILTTLVNPNKKNSYPHYVKSIVASTFIISLFPTTMFMCLDQEVIISNWHWAT TQTTQLSLSFKLDYFSMMFIPVALFVTWSIMEFSLWYMNSDPNINQFFKYLLIFLITMLILVTANNLFQL FIGWEGVGIMSFLLISWWYARADANTAAIQAVLYNRIGDIGFILALAWFILHSNSWDPQQMALLNANPSL TPLLGLLLAAAGKSAQLGLHPWLPSAMEGPTPVSALLHSSTMVVAGIFLLIRFHPLAENSPLIQTLTLCL GAITTLFAAVCALTQNDIKKIVAFSTSSQLGLMMVTIGINQPHLAFLHICTHAFFKAMLFMCSGSIIHNL NNEQDIRKMGGLLKTMPLTSTSLTIGSLALAGMPFLTGFYSKDHIIETANMSYTNAWALSITLIATSLTS AYSTRMILLTLTGQPRFPTLTNINENNPTLLNPIKRLAAGSLFAGFLITNNISPASPFQTTIPLYLKLTA LAVTFLGLLTALDLNYLTNKLKMKSPLCTFYFSNMLGFYPSITHRTIPYLGLLTSQNLPLLLLDLTWLEK LLPKTISQHQISTSIITSTQKGMIKLYFLSFFFPLILTLLLIT 2. Pochodzenie wielorybów W oparciu o podaną sekwencję białka mitochondrialnego określ z jakimi gatunkami lub grupą ssaków najbardziej spokrewniona jest grupa waleni (wielorybów). Przy wyborze sekwencji homologicznych wybierz inne sekwencje należące do waleni, oraz różnych grup ssaków łożyskowych (np. naczelne, parzystokopytne w tym hipopotama, nieparzystokopytne, drapieżne, nietoperze, owadożerne, gryzonie itd.) a także przedstawicieli torbaczy i stekowców (grupę zewnętrzną). gi|5819104|ref|NP_006899.1|ND5_10014 NADH dehydrogenase subunit 5 [Balaenoptera physalus] MNLFTSFTLLTLLILTTPIMMSHTGSHVNNKYQSYVKNIVFCAFITSLVPAMVYLHTNQETLISNWHWIT IQTLKLTLSFKMDYFSLMFMPVALFITWSIMEFSMWYMHSDPYINQFFKYLLLFLITMLILVTANNLFQL FIGWEGVGIMSFLLIGWWFGRTDANTAALQAILYNRIGDIGLLASMAWFLSNMNTWDLEQIFMLNQNPLN FPLMGLVLAAAGKSAQFGLHPWLPSAMEGPTPVSALLHSSTMVVAGIFLLVRFYPLMENNKLIQTVTLCL GAITTLFTAICALTQNDIKKIIAFSTSSQLGLMMVTIGLNQPYLAFLHICTHAFFKAMLFLCSGSIIHNL NNEQDIRKMGGLFKALPFTTTALIIGCLALTGMPFLTGFYSKDPIIEAATSSYTNAWALLLTLIATSLTA VYSTRIIFFALLGQPRFPPSTTINENNPLLINPIKRLLVGSIFAGFILSNSIPPMTTPLMTMPLHLKLTA LAMTTLGFIIAFEINLDTQNLKHKHPSNSFKFSTLLGYFPTIMHRLPPHLDLLMSQKLATSLLDLTWLET ILPKTTALIQLKASTLTSNQQGLIKLYFLSFLITITLSMILFNYPE 1 Analiza filogenetyczna w programie MEGA – sekwencje aminokwasowe 1. Poszukiwanie sekwencji homologicznych algorytmem BLAST. 1.1. Kliknij na ikonę Align i wybierz Do BLAST Serach . 1.2. Wybierz algorytm blastp . 1.3. Wklej w okno Enter Query Sequence sekwencję, do której poszukujesz homologów. 1.4. Jako przeszukiwaną bazę
... zobacz całą notatkę






Komentarze użytkowników (0)